DNA
Implemente um programa que identifique uma pessoa com base em seu DNA, conforme abaixo.
$ python dna.py databases/large.csv sequences/5.txt
Lavender
Começando
Abra o VS Code.
Comece clicando dentro da sua janela de terminal e, em seguida, execute cd por si só. Você deve encontrar que seu "prompt" se parece
com o abaixo.
$
Clique dentro daquela janela de terminal e, em seguida, execute
wget https://cdn.cs50.net/2022/fall/psets/6/dna.zip
seguido de Enter para baixar um ZIP chamado dna.zip
no seu espaço de código. Certifique-se de não ignorar o espaço entre wget e a URL a seguir, ou qualquer outro caractere!
Agora execute
unzip dna.zip
para criar uma pasta chamada dna. Você não precisa
mais do arquivo ZIP, portanto, pode executar
rm dna.zip
e responda com "y" seguido de Enter no prompt para remover o arquivo ZIP que você baixou.
Agora digite
cd dna
seguido de Enter para se mover para dentro desse diretório. Seu prompt deve agora se parecer com o abaixo.
dna/ $
Execute ls sozinho e você deverá ver alguns arquivos e pastas:
databases/ dna.py sequences/Se você tiver algum problema, siga esses mesmos passos novamente e veja se consegue determinar onde errou!
Contexto
O DNA, portador de informações genéticas em seres vivos, tem sido utilizado na justiça criminal por décadas. Mas como exatamente funciona o perfil de DNA? Dada uma sequência de DNA, como os investigadores forenses podem identificar a quem ele pertence?
Bem, o DNA é realmente apenas uma sequência de moléculas chamadas nucleotídeos, dispostas em uma forma específica (uma dupla hélice). Cada célula humana tem bilhões de nucleotídeos dispostos em sequência. Cada nucleotídeo do DNA contém uma das quatro bases diferentes: adenina (A), citosina (C), guanina (G) ou timina (T). Algumas partes dessa sequência (ou seja, genoma) são as mesmas, ou pelo menos muito semelhantes, em quase todos os seres humanos, mas outras partes da sequência apresentam uma diversidade genética maior e, portanto, variam mais na população.
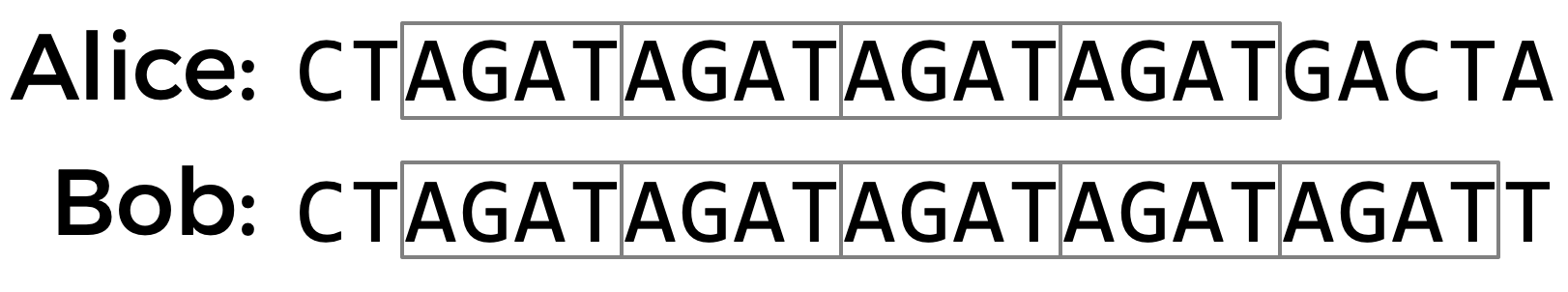
Um local onde o DNA tende a ter alta diversidade genética é em Repetições em Tandem Curtas (STRs). Um STR é uma sequência curta de bases de DNA que tende a se repetir consecutivamente numerosas vezes em locais específicos dentro do DNA de uma pessoa. O número de vezes que um determinado STR se repete varia muito entre os indivíduos. Nas amostras de DNA abaixo, por exemplo, Alice tem o STR AGAT repetido quatro vezes em seu DNA, enquanto Bob tem o mesmo STR repetido cinco vezes.
O uso de vários STRs, em vez de apenas um, pode melhorar a precisão do perfil de DNA. Se a probabilidade de duas pessoas terem o mesmo número de repetições para um único STR é de 5%, e o analista olha para 10 STRs diferentes, então a probabilidade de duas amostras de DNA coincidirem puramente por acaso é de cerca de 1 em 1 quatrilhão (assumindo que todos os STRs são independentes entre si). Portanto, se duas amostras de DNA coincidirem no número de repetições de cada um dos STRs, o analista pode ter bastante confiança de que elas vieram da mesma pessoa. O CODIS, o banco de dados de DNA do FBI, usa 20 STRs diferentes como parte de seu processo de perfil de DNA.
Como seria um banco de dados de DNA desse tipo? Bem, em sua forma mais simples, você pode imaginar a formatação de um banco de dados de DNA como um arquivo CSV, em que cada linha corresponde a um indivíduo e cada coluna corresponde a um STR específico.
name,AGAT,AATG,TATC
Alice,28,42,14
Bob,17,22,19
Charlie,36,18,25
Os dados no arquivo acima sugerem que Alice tem a sequência AGAT repetida 28 vezes consecutivamente em algum lugar do seu DNA, a sequência AATG repetida 42 vezes, e TATC repetida 14 vezes. Enquanto isso, Bob tem esses mesmos três STRs repetidos 17 vezes, 22 vezes e 19 vezes, respectivamente. E Charlie tem esses mesmos três STRs repetidos 36, 18 e 25 vezes, respectivamente.
Então, dada uma sequência de DNA, como você pode identificar a quem ela pertence? Bem, imagine que você procurou na sequência de DNA a sequência consecutiva mais longa de AGATs repetidas e descobriu que a sequência mais longa era de 17 repetições. Se você então descobrisse que a sequência mais longa de AATG é de 22 repetições e a sequência mais longa de TATC é de 19 repetições, isso forneceria uma evidência bastante boa de que o DNA era de Bob. Claro, também é possível que, uma vez que você tenha as contagens para cada um dos STRs, isso não corresponda a ninguém em seu banco de dados de DNA, nesse caso, você não tem uma correspondência.
Na prática, como os analistas sabem em qual cromossomo e em qual localização do DNA um STR será encontrado, eles podem localizar sua pesquisa apenas em uma seção estreita do DNA. Mas vamos ignorar esse detalhe para este problema.
Sua tarefa é escrever um programa que receberá uma sequência de DNA e um arquivo CSV contendo contagens de STRs para uma lista de indivíduos e, em seguida, indicar a quem o DNA (provavelmente) pertence.
Especificação
Em um arquivo chamado dna.py, implemente um programa que identifique a quem pertence uma sequência de DNA.
- O programa deve exigir como seu primeiro argumento de linha de comando o nome de um arquivo CSV contendo as contagens de STR para uma lista de indivíduos e deve exigir como seu segundo argumento de linha de comando o nome de um arquivo de texto contendo a sequência de DNA a ser identificada.
- Se o programa for executado com o número incorreto de argumentos de linha de comando, o programa deve imprimir uma mensagem de erro de sua escolha (com
print). Se o número correto de argumentos for fornecido, você pode assumir que o primeiro argumento é de fato o nome de arquivo de um arquivo CSV válido e que o segundo argumento é o nome de arquivo de um arquivo de texto válido.
- Se o programa for executado com o número incorreto de argumentos de linha de comando, o programa deve imprimir uma mensagem de erro de sua escolha (com
- Seu programa deve abrir o arquivo CSV e ler seu conteúdo na memória.
- Você pode assumir que a primeira linha do arquivo CSV será os nomes das colunas. A primeira coluna será a palavra
namee as colunas restantes serão as sequências de STR em si.
- Você pode assumir que a primeira linha do arquivo CSV será os nomes das colunas. A primeira coluna será a palavra
- Seu programa deve abrir a sequência de DNA e ler seu conteúdo na memória.
- Para cada um dos STRs (da primeira linha do arquivo CSV), seu programa deve calcular a maior sequência de repetições consecutivas do STR na sequência de DNA para identificar. Observe que definimos uma função auxiliar para você,
longest_match, que fará exatamente isso! - Se as contagens do STR corresponderem exatamente a qualquer um dos indivíduos no arquivo CSV, seu programa deve imprimir o nome do indivíduo correspondente.
- Você pode assumir que as contagens do STR não corresponderão a mais de um indivíduo.
- Se as contagens do STR não corresponderem exatamente a nenhum dos indivíduos no arquivo CSV, seu programa deve imprimir
No match.
Uso
Seu programa deve se comportar conforme o exemplo abaixo:
$ python dna.py databases/large.csv sequences/5.txt
Lavender
$ python dna.py
Usage: python dna.py data.csv sequence.txt
$ python dna.py data.csv
Usage: python dna.py data.csv sequence.txt
Dicas
- Você pode achar o módulo
csvdo Python útil para ler arquivos CSV em memória. Você pode querer aproveitar tanto ocsv.readerquanto ocsv.DictReader. - As funções
openereadpodem ser úteis para ler arquivos de texto em memória. - Considere quais estruturas de dados podem ser úteis para manter o controle das informações em seu programa. Uma
listaou umdicionáriopodem ser úteis. - Lembre-se que definimos uma função (
longest_match) que, dada uma sequência de DNA e um STR como entrada, retorna o número máximo de vezes que o STR se repete. Você pode então usar essa função em outras partes do seu programa!
Testando
Embora o check50 esteja disponível para este problema, você é incentivado a testar seu código primeiro por conta própria para cada um dos seguintes testes.
- Execute seu programa como
python dna.py databases/small.csv sequences/1.txt. Seu programa deve exibirBob. - Execute seu programa como
python dna.py databases/small.csv sequences/2.txt. Seu programa deve exibirNo match. - Execute seu programa como
python dna.py databases/small.csv sequences/3.txt. Seu programa deve exibirNo match. - Execute seu programa como
python dna.py databases/small.csv sequences/4.txt. Seu programa deve exibirAlice. - Execute seu programa como
python dna.py databases/large.csv sequences/5.txt. Seu programa deve exibirLavender. - Execute seu programa como
python dna.py databases/large.csv sequences/6.txt. Seu programa deve exibirLuna. - Execute seu programa como
python dna.py databases/large.csv sequences/7.txt. Seu programa deve exibirRon. - Execute seu programa como
python dna.py databases/large.csv sequences/8.txt. Seu programa deve exibirGinny. - Execute seu programa como
python dna.py databases/large.csv sequences/9.txt. Seu programa deve exibirDraco. - Execute seu programa como
python dna.py databases/large.csv sequences/10.txt. Seu programa deve exibirAlbus. - Execute seu programa como
python dna.py databases/large.csv sequences/11.txt. Seu programa deve exibirHermione. - Execute seu programa como
python dna.py databases/large.csv sequences/12.txt. Seu programa deve exibirLily. - Execute seu programa como
python dna.py databases/large.csv sequences/13.txt. Seu programa deve exibirNenhuma correspondência. - Execute seu programa como
python dna.py databases/large.csv sequences/14.txt. Seu programa deve exibirSeverus. - Execute seu programa como
python dna.py databases/large.csv sequences/15.txt. Seu programa deve retornarSirius. - Execute seu programa como
python dna.py databases/large.csv sequences/16.txt. Seu programa deve retornarNo match. - Execute seu programa como
python dna.py databases/large.csv sequences/17.txt. Seu programa deve retornarHarry. - Execute seu programa como
python dna.py databases/large.csv sequences/18.txt. Seu programa deve retornarNo match. - Execute seu programa como
python dna.py databases/large.csv sequences/19.txt. Seu programa deve retornarFred. - Execute seu programa como
python dna.py databases/large.csv sequences/20.txt. Seu programa deve retornarNo match.
Execute o código abaixo para avaliar a correção do seu código usando o check50. Mas certifique-se de compilá-lo e testá-lo por conta própria também!
check50 cs50/problems/2023/x/dnaExecute o código abaixo para avaliar o estilo do seu código usando style50.
style50 dna.pyComo Enviar
No seu terminal, execute o seguinte comando para enviar o seu trabalho.
submit50 cs50/problems/2023/x/dna